Noticias
Proteínas humanas: su mapa ya está muy cerca

Las proteínas humanas (bueno, las proteínas en general) son fascinantes. Y sí, sé que esa frase ha sonado a snob amante de la ciencia y de los documentales de La 2, pero es que es cierto. Su estructura es un enorme misterio para investigadores de todo el mundo, y de su comprensión dependen medicina, biología, farmacia… llegar a crear un mapa que reúna todas ellas y nos permita conocer su morfología puede suponer una revolución en todos estos campos.
El problema es que sus estructuras son bastante complejas, con multitud de pliegues, lo que da lugar a formas absolutamente irregulares, algo que dificulta sustancialmente la posibilidad de analizarlas. Y lo que hace clave que sea tan interesante el poder clasificar y mapear todas las proteínas humanas, es que su función viene determinada por su forma. Y cuando digo que sus formas son complejas, me refiero a que son realmente complejas:
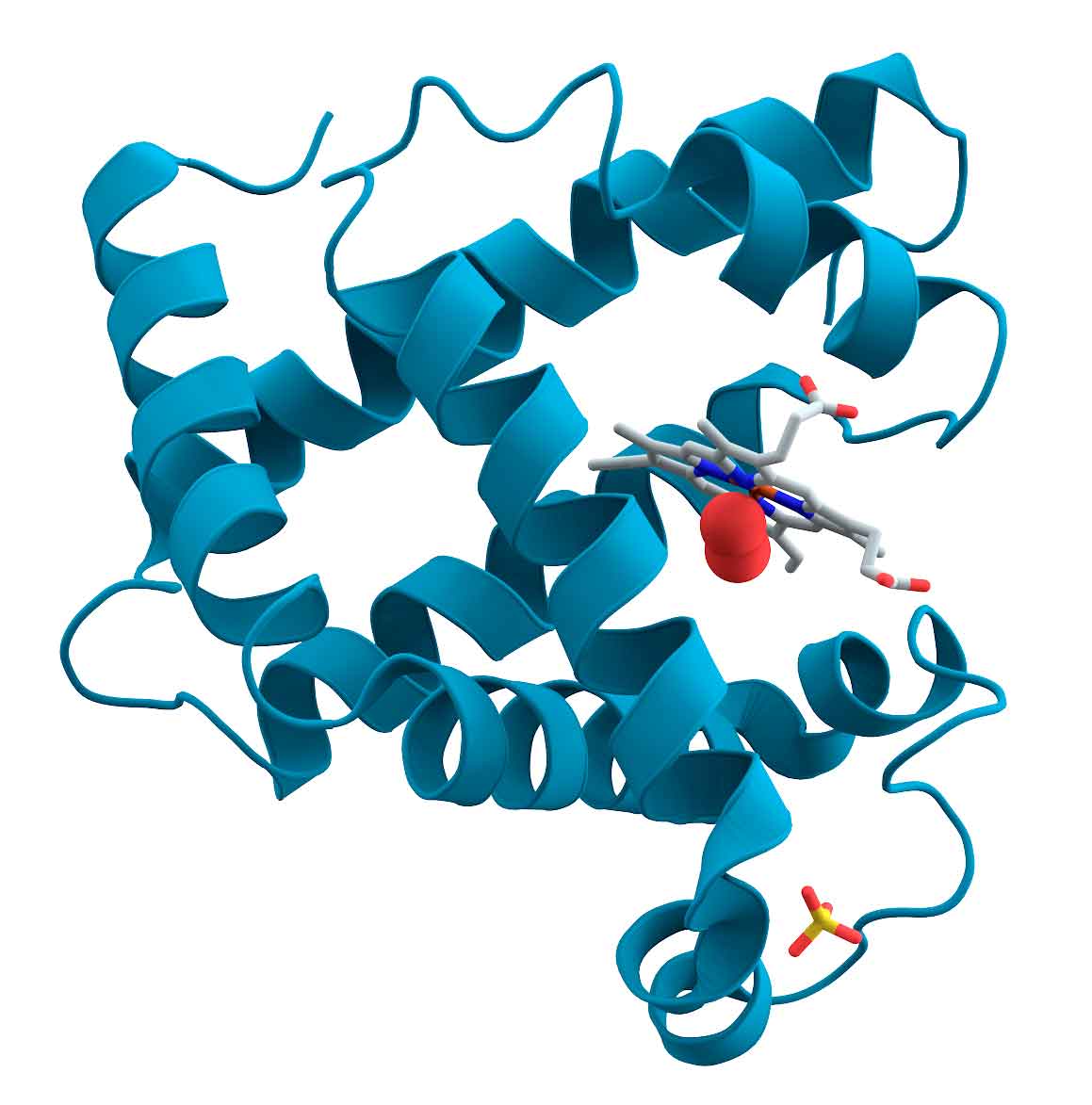
Representación tridimensional de la mioglobina. Imagen: AzaToth
Como puedes imaginar al ver su aspecto y tener en cuenta su tamaño, es fácilmente deducible la enorme complejidad que plantea analizar cada una de ellas. Y si existieran unos centenares o incluso unos miles de tipos, trazar ese mapa sería enormemente complejo, pero factible. El problema es que a esta dificultad debemos sumar que existen varios cientos de miles de proteínas humanas. Abordar este estudio queda, por lo tanto, totalmente fuera de alcance.
Sin embargo, y como ya he dicho anteriormente, esta imposibilidad se circunscribe a los medios convencionales para realizar este tipo de análisis, pero en esta ocasión la ciencia ha vuelto a contar con la colaboración de una tecnología muy relacionada con la investigación: la inteligencia artificial. Y es que haciendo uso de la misma, DeepMind ha logrado crear el mapa de proteínas humanas más grande trazado hasta el momento, con más de 360.000 formas.
No es este el primer gran logro de DeepMind en relación con las proteínas humanas. Ya el año pasado lograron desentrañar el modo en el que éstas adquieren su morfología, un elemento determinante en cómo encajan entre sí. Tras ese logro, la compañía anunció AlphaFold 2, un proyecto para trazar el mapa de las proteínas humanas mediante predicción por parte de una IA entrenada por la compañía. Poco más de medio año después, el proyecto se ha completado y, además, DeepMind va a poner este mapa a la disposición de todo el mundo, haciéndolo público y gratuito.
Este mapa, eso sí, se basa en predicciones desarrolladas por la IA de la compañía, por lo que su nivel de fiabilidad no llega al de los perfiles trazados por los medios clásicos. Sin embargo, tras comprobar las similitudes de proteínas humanas ya estudiadas y las estimaciones realizadas por la IA, parece probado que los resultados que ofrece este sistema son altamente fiables, por lo que proporcionan un nivel de conocimiento único e impensable hace tan solo un año.
De las proteínas humanas depende desde la lucha contra las enfermedades hasta la creación de tejidos. Es por ello que la comprensión sobre los pliegues de las mismas y el modo en el que se combinan entre sí puede ser clave en la biología y la medicina del siglo XXI, a un nivel que podemos comparar con el de la secuenciación del genoma humano, una tarea que, en una sorprendente coincidencia, se completó hace solo unas semanas, pues hasta ahora todavía quedaban algunos pares por definir.
Así pues, en poco más de un mes se ha completado el mapa del genoma humano y se ha establecido el proteoma humano, dos pasos de gigante. Es cierto que el segundo, el de las proteínas humanas está basado en predicciones, pero supone un excelente primer paso, que sin duda alguna estimulará miles de nuevas investigaciones que se basarán en estos datos. Un día fabuloso para la ciencia.
Más información: DeepMind
-

 GuíasHace 6 días
GuíasHace 6 díasQué placa base elegir: Guía de compras para Intel y AMD
-

 GuíasHace 2 días
GuíasHace 2 díasGuía para diferenciar generaciones y gamas de tarjetas gráficas
-

 A FondoHace 6 días
A FondoHace 6 díasOfertas Flash del Black Friday 2024 de PcComponentes
-

 A FondoHace 4 días
A FondoHace 4 díasGeForce RTX 5050, especificaciones, rendimiento, equivalencias, fecha de lanzamiento y posible precio























